小分子机器学习模型的训练数据覆盖偏差问题研究

小分子机器学习旨在预测分子的化学或生物特性,广泛应用于毒性预测、药物研发等领域。近年来,端到端模型备受关注,但这些模型依赖于训练数据的代表性,忽略了数据覆盖偏差可能带来的风险。
德国一个研究团队近期发表在《Nature Communications》上的论文(Coverage bias in small molecule machine learning),深入探讨了大规模数据集对已知生物分子结构空间的覆盖情况。他们提出了一种基于最大公共边子图 (MCES) 的距离度量方法,更准确地评估分子结构间的相似性,并以此分析了常用数据集的覆盖偏差。

研究结果显示,许多常用数据集缺乏对生物分子结构的全面覆盖,这限制了模型的预测能力。研究人员提出了两种补充方法来评估训练数据集与已知分子分布的差异,为未来数据集构建提供了指导。
数据集分析方法
该研究团队采用了一种基于最大公共边子图 (MCES) 的新距离度量方法,克服了传统分子指纹方法的不足,更有效地捕捉分子结构的相似性。他们利用均匀流形近似和投影 (UMAP) 技术对生物分子结构进行可视化,直观地展现了数据集的覆盖情况。
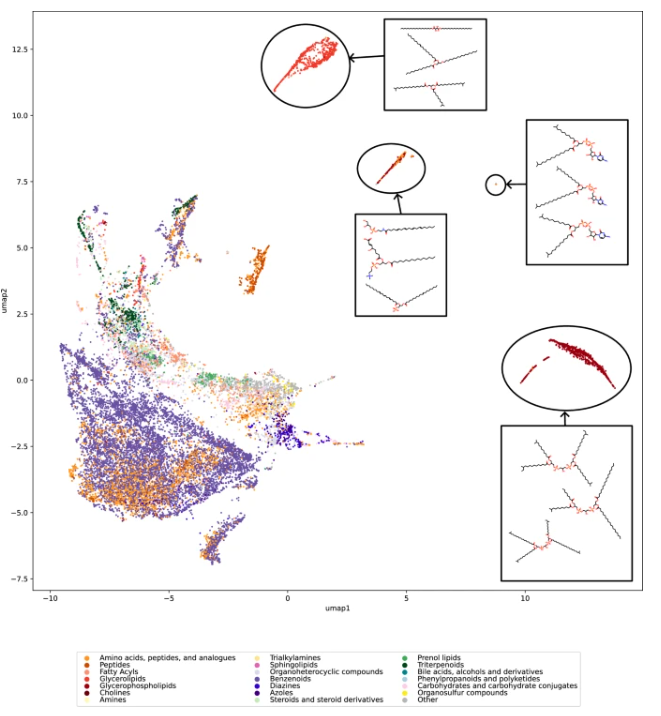
图示:生物分子结构的初始图谱 (论文截图)
研究人员对 20,000 个生物分子结构进行了抽样分析,并对 10 个常用的公共数据集进行了评估。结果表明,这些数据集的分子结构分布通常远非均匀,许多生物分子结构区域存在空白。
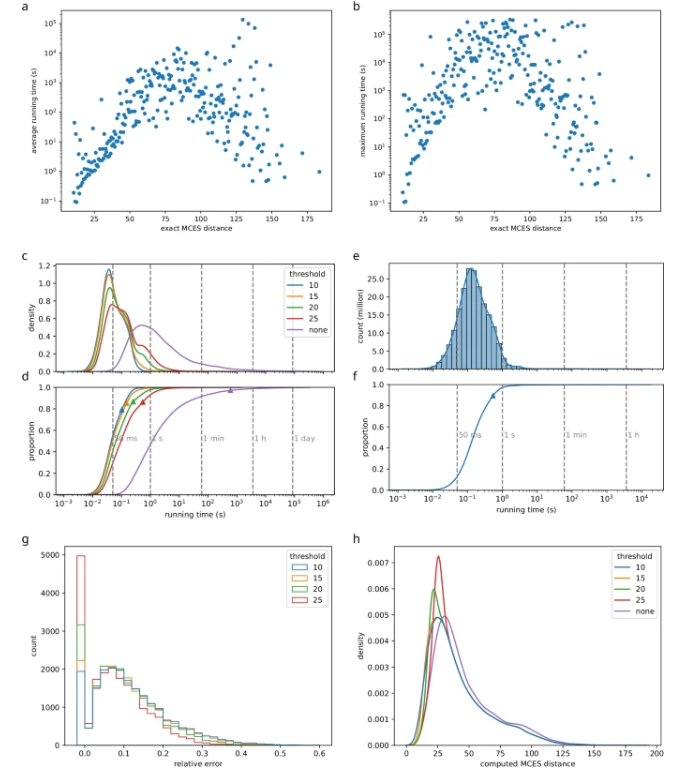
图示:最大公共边子图计算 (论文截图)
结论与展望
该研究揭示了小分子机器学习中训练数据覆盖偏差的普遍性及其对模型性能的负面影响。研究人员强调,构建具有代表性的数据集至关重要,并建议将数据集分布分析纳入机器学习最佳实践指南中。 他们提出的 MCES 距离度量方法为评估数据集覆盖情况提供了一种有效工具,有助于改进模型的泛化能力和预测精度。 未来研究可进一步优化MCES计算效率,并将其集成到机器学习模型的训练流程中。











